Contaminant monitoring and detection of alkB genes in the Argentine-Uruguayan Common Fishing Zone
DOI:
https://doi.org/10.47193/mafis.3612023010109Keywords:
Marine environment, persistent contaminants, catabolic genesAbstract
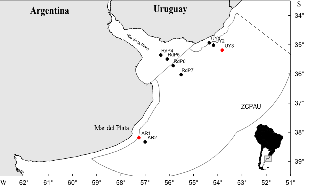
In this study, the presence of diverse pollutants in sediments samples was determined in the Río de la Plata and the Argentine-Uruguayan Common Fishing Zone (AUCFZ 35° S-38° S). In addition, the occurrence of microbial genes alkB, used as functional markers, was analyzed in order to determine the hydrocarbon degradation potential along this marine environment. Heavy metals were detected in sampling sites UY1, RdP4 and AR2. Cr was found in a range from < 5.0 to 20.7 mg kg-1, and Pb from not detectable (ND) to 26.0 mg kg-1. Both Cd and Hg showed values under detection limits (< 0.2 mg kg-1 and < 0.01 mg kg-1, respectively). Cu varied between ND and 24.6 mg kg-1, and it was the only metal that overcame the values recommended by the ISQG (Interim Sediment Quality Guideline), in the guide levels for biota protection in sediments (CCME, Canadian Council of Ministers of the Environment). Hydrocarbons, atrazine, glyphosate + AMPA (aminomethyl phosphonic acid) and pesticides values were under the quantification limits, while PCBs (from < 20.0 to 77.7 µg kg-1) exceeded the action level A for the ‘Recommendations for the Management of Dredging Material in Spanish Ports’. Purified microbial genomic DNA was obtained in eight from the nine analyzed samples, and the catabolic gen alkB was amplified in sampling sites UY2, UY1, RdP4, AR2 and AR1. Further studies are needed to evaluate the microbial biodegradation potential in this area. These researches mean a valuable input in order to investigate the impact of the anthropogenic disturbances on marine ecosystems and to understand the mechanisms of natural attenuation.
Downloads
References
Acha EM, Mianzan H, Guerrero R, Carreto J, Giberto D, Montoya N, Carignan M. 2008. An overview of physical and ecological processes in the Río de la Plata Estuary. Cont Shel Res. 28: 1579-1588.
Buceta JL, Lloret A, Antequera M, Obispo R, Sierra J, Martínez-Gil M. 2015. Nuevo marco para la caracterización y clasificación del material dragado en España. Ribagua, 2 (2): 105-115.
Cappelletti N, Migoya C, Skorupka CN, Toranzo S, Lasci J, Colombo JC. 2006. Hidrocarburos alifáticos, aromáticos, hopanos y PCBs en sedimentos superficiales de la zona de máxima turbidez del Río de la Plata. En: VI Jornadas Nacionales de Ciencias del Mar, Puerto Madryn, Argentina. p. 13.
Carsen AE, Perdomo A, Arriola M. 2005. Contaminación de aguas, sedimentos y biota. En: Documento Técnico, Proyecto PNUD/GEF RLA/99/G31 “Protección Ambiental del Río de la Plata y su Frente Marítimo: Prevención y Control de la Contaminación y Restauración de Hábitats” FREPLATA. Montevideo: Fondo para el Medio Ambiente Mundial (FMAM), Programa de las Naciones Unidas para el Desarrollo (PNUD). p. 57-88.
Catania V, Cappello S, Di Giorgi V, Santisi S, Di Maria R, Mazzola A, Vizzini S, Quatrini P. 2018. Microbial communities of polluted sub-surface marine sediments. Mar Pollut Bull. 131: 396-406.
[CCME] Canadian Council of Ministers of the Environment. 2001. Canadian Environmental Quality Guidelines (CEQGs) provide science-based goals for the quality of aquatic and terrestrial ecosystems, summary table. CCME. [accessed 2022 March]. https://ccme.ca/en/summary-table.
[CEDEX] Centro de Estudios y Experimentación de Obras Públicas. 1994. Recomendaciones para la gestión del material dragado en los puertos españoles. Madrid: Ministerio de Obras Públicas, Transportes y Medio Ambiente. 45 p.
Colombo JC, Barreda A, Bilos C, Cappelletti N, Demichelis S, Lombardi P, Migoya MC, Skorupka CN, Suarez G. 2004. Oil spill in the Río de la Plata estuary, Argentina: 1. Biogeochemical assessment of waters, sediments, soils and biota. Environ Pollut. 134: 277-289.
Colombo JC, Barreda A, Bilos C, Cappelletti N, Migoya MC, Skorupka CN. 2005. Oil spill in the Río de la Plata estuary, Argentina: 2-hydrocarbon disappearance rates in sediments and soils. Environ Pollut. 134: 267-276.
Colombo JC, Bilos C, Remes Lenicov M, Colautti D, Landoni P, Brochu C. 2000. Detritivorous fish contamination in the Río de la Plata estuary. A critical accumulation pathway in the cycle of anthropogenic compounds. Can J Fish Aquat Sci. 57: 1139-1150.
Colombo JC, Skorupka CN. Cappelletti N, Migoya MC, Toranzo S, Lasci J, Tatone LM. 2006. Composición elemental y contaminantes persistentes en sedimentos de la zona de máxima turbidez del Río de la Plata. VI Jornadas Nacionales de Ciencias del Mar, Puerto Madryn, Argentina. p. 33.
Corti-Monzon G, Nisenbaum M, Peressutti S, Junca H, Garcia-Bonilla E, Murialdo SE. 2021. Oily Bilge Wastes Harbor a Set of Persistent Hydrocarbonoclastic Bacteria Accompanied by a Variable alkB Gene Composition in Marine Vessel Samples from Southwestern Atlantic Port of Mar del Plata, Argentina. Water Air Soil Pollut. 232: 301.
Díaz Jaramillo M, Gonzalez M, Pegoraro CA. 2018. Distribución vertical de hidrocarburos aromáticos policíclicos (HAPs) en sedimentos estuarinos de relevancia biológica. X Jornadas Nacionales de Ciencias del Mar, Buenos Aires, Argentina. p. 410.
Djahnit N, Chernai S, Catania V, Hamdi B, China B, Cappello S, Quatrini P. 2019. Isolation, characterization and determination of biotechnological potential of oil-degrading bacteria from Algerian centre coast. J Appl Microbiol. 126: 780-795.
Elliott M, Quintino V. 2007. The Estuarine Quality Paradox, environmental homeostasis and the difficulty of detecting anthropogenic stress in naturally stressed areas. Mar Pollut Bull. 54: 640-605.
Ellis RJ, Morgan P, Weightman AJ, Fry JC. 2003. Cultivation-dependent and -independent approaches for determining bacterial diversity in heavy-metal-contaminated soil. Appl Environ Microbiol. 69: 3223-3230.
[EPA] United States Environmental Protection Agency. 2001. Methods for collection, storage, and manipulation of sediments for chemical and toxicological analyses technical manual (EPA 823-B-01-002).
Ernst F, Alonso B, Colazzo M, Pareja L, Cesio V, Pereira A, Márquez A, Errico E, Segura AM, Heinzen H, Pérez-Parada A. 2018. Occurrence of pesticide residues in fish from South American rainfed agroecosystems. Sci Total Environ. 631-632: 169-179.
Guibert LM, Loviso CL, Marcos MS, Commendatore MG, Dionisi HM, Lozada M. 2012. Alkane biodegradation genes from chronically polluted subantarctic coastal sediments and their shifts in response to oil exposure. Microb Ecol. 64: 605-616.
Heiss-Blanquet S, Benoit Y, Maréchaux C, Monot F. 2005. Assessing the role of alkane hydroxylase genotypes in environmental samples by competitive PCR. J Appl Microbiol. 99: 1392-1403.
Iwai S, Johnson TA, Chai B, Hashsham SA, Tiedje JM. 2011. Comparison of the specificities and efficacies of primers for aromatic dioxygenase gene analysis of environmental samples. Appl Environ Microbiol. 77: 3551-3557.
Izzo SA, Quintana S, Espinosa M, Babay PA, Peressutti SR. 2018. First characterization of PAH degrading bacteria from Río de la Plata and high resolution melting: an encouraging step towards bioremediation. Environ Technol. 40: 1250-1261.
Jaureguizar AJ, Menni RC, Bremec CS, Mianzan HW, Lasta CA. 2003. Fish assemblage and environmental patterns in the Río de la Plata estuary. Estuar Coast Shelf Sci. 56: 921-933.
Kloos K, Munch JC, Schloter M. 2006. A new method for the detection of alkane monooxygenase homologous genes (alkB) in soils based on PCR-hybridization. J Microbiol Methods. 66: 486-496.
Leahy JG, Colwell RR. 1990. Microbial degradation of hydrocarbons in the environment. Microbiol Rev. 54: 305-315.
Li H, Wang XL, Mu BZ, Gu JD, Liu YD, Lin KF, Lu SG, Lu Q, Li BZ, Li Y, Du XM. 2013. Molecular detection, quantification and distribution of alkane-degrading bacteria in production water from low temperature oilfields. Int Biodeterior Biodegrad. 76: 49-57.
Marcos MS, Lozada M, Di Marzio WD, Dionisi HM. 2012. Abundance, dynamics, and biogeographic distribution of seven polycyclic aromatic hydrocarbon dioxygenase gene variants in coastal sediments of Patagonia. Appl Environ Microbiol. 78: 1589-1592.
McGenity TJ, Folwell BD, Mckew BA, Sanni GO. 2012. Marine crude-oil biodegradation: a central role for interspecies interactions. Aquat Biosyst. 8: 1.
Mianzan HW, Lasta C, Acha EM, Guerrero R, Macchi G, Bremec C. 2001. The Río de la Plata estuary, Argentina-Uruguay. En: Seeliger U, De Lacerda LD, editores. Ecological studies: coastal marine ecosystems of Latin America. Berlin: Springer-Verlag. p. 185-204.
Militelli MI, Rodriguez KA, Cortés F, Macchi GJ. 2013. Influence of environmental factors on spawning of sciaenids from the Buenos Aires Coastal Zone, Argentina. Cienc Mar. 39: 55-68.
Morales-Caselles C, Rica A, Abbondanzi F, Campisi T, Iacondini A, Riba I, Delvalls A. 2008. Assessing sediment quality in Spanish ports using a green algae bioassay. Cienc Mar. 34: 329-337.
Nie Y, Chi CQ, Fang H, Liang JL, Lu SL, Lai GL, Tang YQ, Wu XL. 2014. Diverse alkane hydroxylase genes in microorganisms and environments. Sci Rep. 4: 1-11.
Olivera NL, Nievas ML, Lozada M, Del Prado G, Dionisi HM, Simeriz F. 2009. Isolation and characterization of biosurfactant-producing Alcanivorax strains: hydrocarbon accession strategies and alkane hydroxylase gene analysis. Res Microbiol. 160: 19-26.
Purohit HJ, Kapley A, Moharikar AA, Narde G. 2003. A novel approach for extraction of PCR-compatible DNA from activated sludge samples collected from different biological effluent treatment plants. J Microbiol Methods. 52: 315-323.
Salomons W, Brils J. 2004. Contaminated sediments in european River Basins. Eur Sedim Res Net. 1: 1-79.
Sharma N, Tanksale H, Kapley A, Purohit HJ. 2012. Mining the metagenome of activated biomass of an industrial wastewater treatment plant by a novel method. Indian J Microbiol. 52: 538-543.
Smith CB, Tolar BB, Hollibaugh JT, King GM. 2013. Alkane hydroxylase gene (alkB) phylotype composition and diversity in northern Gulf of Mexico bacterioplankton. Front Microbiol. 4: 370.
Tatone LM, Bilos C, Skorupka, CN, Toranzo S, Colombo JC. 2006. Metales pesados en sedimentos superficciales de la zona de máxima turbidez del Río de la Plata. VI Jornadas Nacionales de Ciencias del Mar. Puerto Madryn, Argentina. p. 344.
Venturini N, Bícego MC, Taniguchi S, Sasaki ST, García-Rodríguez F, Brugnoli E, Muniz P. 2015. A multi-molecular marker assessment of organic pollution in shore sediments from the Río de la Plata estuary, SW Atlantic. Mar Pollut Bull. 91: 461-475.
Viggor S, Juhanson J, Joesaar M, Mitt M, Truu J, Vedler E, Heinaru A. 2013. Dynamic changes in the structure of microbial communities in Baltic Sea coastal seawater microcosms modified by crude oil, shale oil or diesel fuel. Microbiol Res. 168: 415-427.
Wang S, Li G, Liao Z, Liu T, Ma T. 2022. A novel alkane monooxygenase (alkB) clade revealed by massive genomic survey and its dissemination association with IS elements. PeerJ. 10: e14147.
Wang W, Shao Z. 2012. Diversity of flavin-binding monooxygenase genes (almA) in marine bacteria capable of degradation long-chain alkanes. FEMS Microbiol Ecol. 80: 523-533.
White JC, Triplett T. 2002. Polycyclic aromatic hydrocarbons (PAHs) in the sediments and fish of the Mill River, New Haven, Connecticut, USA. Bull Environ Contam Toxicol. 68: 104-110.
Whyte LG, Smits TH, Labbe D, Witholt B, Greer CW, Van Beilen JB. 2002. Gene cloning and characterization of multiple alkane hydroxylase systems in Rhodococcus strains Q15 and NRRL B-16531. Appl Environ Microbiol. 68: 5933-5942.
Downloads
Published
Issue
Section
License
Copyright (c) 2023 Silvia R. Peressutti, Pablo A. Zorzoli

This work is licensed under a Creative Commons Attribution-NonCommercial-ShareAlike 4.0 International License.
Authors of articles published in Marine and Fishery Sciences retain copyright on their articles, except for any third-party images and other materials added by Marine and Fishery Sciences, which are subject to copyright of their respective owners. Authors are therefore free to disseminate and re-publish their articles, subject to any requirements of third-party copyright owners and subject to the original publication being fully cited. Visitors may also download and forward articles subject to the citation requirements. The ability to copy, download, forward or otherwise distribute any materials is always subject to any copyright notices displayed. Copyright notices must be displayed prominently and may not be obliterated, deleted or hidden, totally or partially.
This journal offers authors an Open Access policy. Users are allowed to read, download, copy, distribute, print, search, or link to the full texts of the articles, or use them for any other legal purpose within the Creative Commons 4.0 license (BY-NC-SA), without asking prior permission from the publisher or the author. This is in accordance with the BOAI definition of Open Access.