Cerrando la brecha genómica: la necesidad de una genómica de peces coordinada en Argentina
DOI:
https://doi.org/10.47193/mafis.3912026010111Palabras clave:
Genética de la conservación, genómica en acuicultura, diversidad genética, pesquerías, genomaResumen
La genómica de peces se ha consolidado como una herramienta clave para comprender los procesos biológicos fundamentales, como la biodiversidad, la evolución, la adaptación, pero también en aplicaciones directas en conservación, acuicultura y manejo pesquero. Argentina, con más de 1.100 especies de peces y una gran diversidad de ecosistemas acuáticos, presenta un enorme potencial para el desarrollo de estudios genómicos. Aunque históricamente subrepresentado en las bases de datos globales, el país ha comenzado a generar avances significativos (incluyendo la secuenciación de especies de importancia comercial y ecológica). Esta nota resume el estado actual de la genómica de peces en Argentina, destaca aportes relevantes y enfatiza la necesidad de promover ensamblajes genómicos de calidad, esfuerzos nacionales coordinados y una mayor cobertura taxonómica para preservar adecuadamente la biodiversidad acuática del país.
Descargas
Referencias
Bauni V, Bertonatti C, Giacchino A, Schivo F, Mabragaña E, Roesler I, Rosso JJ, Teta P, Williams JD, Abba AM, et al. 2022. Biodiversity of vertebrates in Argentina: patterns of richness, endemism and conservation status. ZooKeys. 1085: 101-127. DOI: https://doi.org/10.3897/zookeys.1085.76033 DOI: https://doi.org/10.3897/zookeys.1085.76033
Bista I, McCarthy SA, Wood J, Ning Z, Detrich III HW, Desvignes T, Postlethwait J, Chow W, Howe K, Torrance J, et al. 2020. The genome sequence of the channel bull blenny, Cottoperca gobio (Günther, 1861). Wellcome Open Res. 5: 148. DOI: https://doi.org/10.12688/wellcomeopenres.16012.1 DOI: https://doi.org/10.12688/wellcomeopenres.16012.1
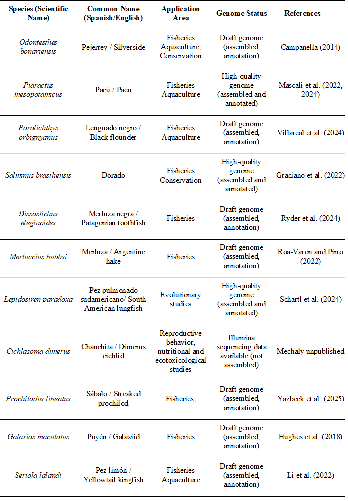
Campanella D. 2014. Evolutionary history and whole genome sequence of pejerrey (Odontesthes bonariensis): new insights into sex determination in fishes [PhD thesis]. Washington: The George Washington University. https://scholarspace.library.gwu.edu/etd/mp48sd085.
Cheng CHC, Rivera-Colón AG, Minhas BF, Wilson L, Rayamajhi N, Vargas-Chacoff L, Catchen JM. 2023. Chromosome-level genome assembly and circadian gene repertoire of the Patagonia blennie Eleginops maclovinus, the closest ancestral proxy of Antarctic Cryonotothenioids. Genes. 14: 1196. DOI: https://doi.org/10.3390/genes14061196 DOI: https://doi.org/10.3390/genes14061196
Fan G, Song Y, Yang L, Huang X, Zhang S, Zhang M, Yang X, Chang Y, Zhang H, Li Y, et al. 2020. Initial data release and announcement of the 10,000 Fish Genomes Project (Fish10K). Gigascience. 9 (8): 80. DOI: https://doi.org/10.1093/gigascience/giaa080 DOI: https://doi.org/10.1093/gigascience/giaa080
Fricke R, Eschmeyer WN, Van der Laan R, editors. 2025. Eschmeyer’s catalog of fishes: genera, species, references. http://researcharchive.calacademy.org/research/ichthyology/catalog/fishcatmain.asp.
Cardoso YP, Brancolini F, Paracampo AH, Lizarralde MS, Covain R, Montoya-Burgos JI. 2016. Hypostomus formosae, a new catfish species from the Paraguay river basin with redescription of H. boulengeri (Siluriformes: Loricariidae). Ichthyol Explor Freshw. 27: 9-23.
Cardoso YP, Brancolini F, Protogino L, Paracampo A, Bogan S, Posadas P, Montoya-Burgos JI. 2019. An integrated approach clarifies the cryptic diversity in Hypostomus Lacépède 1803 from the Lower La Plata Basin. An Acad Bras Cienc. 91 (2): e20180131. DOI: https://doi.org/10.1590/0001-3765201920180131 DOI: https://doi.org/10.1590/0001-3765201920180131
Graciano RCD, Oliveira RS, Santos IM, Yazbeck GM. 2022. Genomic resources for Salminus brasiliensis. Front Genet. 13: 855718. DOI: https://doi.org/10.3389/fgene.2022.855718 DOI: https://doi.org/10.3389/fgene.2022.855718
Hirt-Chabbert JA, Mechaly AS, Tapia C. 2024. Seafood in Argentina: marine fish species, seasonal occurrence and prices. Rev Fish Biol Fish. 34 (2): 753-774. DOI: https://doi.org/10.1007/s11160-024-09836-4 DOI: https://doi.org/10.1007/s11160-024-09836-4
Hughes LC, Ortí G, Huang Y, Sun Y, Baldwin CC, Thompson AW, Arcila D, Betancur-R R, Li C, Becker L, et al. 2018. Comprehensive phylogeny of ray-finned fishes (Actinopterygii) based on transcriptomic and genomic data. Proc Natl Acad Sci USA. 115 (24): 6249-6254. DOI: https://doi.org/10.1073/pnas.1719358115 DOI: https://doi.org/10.1073/pnas.1719358115
Li S, Liu K, Cui A, Hao X, Wang B, Wang H-Y, Jiang Y, Wang Q, Feng B, Xu Y, et al. 2022. Chromosome-level genome assembly of yellowtail kingfish (Seriola lalandi). Front Genet. 12: 825742. DOI: https://doi.org/10.3389/fgene.2021.825742 DOI: https://doi.org/10.3389/fgene.2021.825742
Lu G, Luo M. 2020. Genomes of major fishes in world fisheries and aquaculture: status, application and perspective. Aquac Fish. 5 (4): 163-173. DOI: https://doi.org/10.1016/j.aaf.2020.05.004 DOI: https://doi.org/10.1016/j.aaf.2020.05.004
Mascali FC, Mastrochirico-Filho VA, Posner VM, Rubiolo JA, Hashimoto DT, Villanova GV. 2024. High-quality genome assembly and annotation of the pacu Piaractus mesopotamicus: an aquatic genetic resource of South American aquaculture. Aquacult Int. 32: 8981-9003. DOI: https://doi.org/10.1007/s10499-024-01601-5 DOI: https://doi.org/10.1007/s10499-024-01601-5
Mascali FC, Posner VM, Romero Marano EA, del Pazo F, Hermida M, Sánchez S, Mazzoni TS, Martinez P, Rubiolo JA, Villanova GV. 2022. Development and validation of sex-specific markers in Piaractus mesopotamicus. Aquaculture. 558. DOI: https://doi.org/10.1016/j.aquaculture.2022.738374 DOI: https://doi.org/10.1016/j.aquaculture.2022.738374
Mechaly AS, Awruch C, Cabrita E, Costas B, Fernandes JMO, Gallego V, Hirt- Chabbert J, Konstantinidis I, Olivera C, Ramos-Júdez S, et al. 2024. Cutting-edge methods in teleost and chondrichthyan reproductive biology. Rev Fish Sci Aquac. 33 (1): 77-112. DOI: https://doi.org/10.1080/23308249.2024.2377999 DOI: https://doi.org/10.1080/23308249.2024.2377999
Mechaly AS, Elizur A, Escobar-Aguirre S, Hirt-Chabbert J, Inohuye Rivera RB, Knibb W, Nocillado J, Pérez-Urbiola JC, Premachandra HKA, Somoza GM, et al. 2025. Advances in reproduction, nutrition and disease research in the yellowtail kingfish (Seriola lalandi) aquaculture. Rev Aqua. 17: e70034. DOI: https://doi.org/10.1111/raq.70034 DOI: https://doi.org/10.1111/raq.70034
Rhie A, McCarthy SA, Fedrigo O, Damas J, Formenti G, Koren S, Uliano-Silva M, Chow W, Fungtammasan A, Kim J, et al. 2021. Towards complete and error-free genome assemblies of all vertebrate species. Nature. 592: 737-746. DOI: https://doi.org/10.1038/s41586-021-03451-0 DOI: https://doi.org/10.1038/s41586-021-03451-0
Roa-Varon A, Pirro S. 2022. The complete genome sequences of 18 species of Gadiformes (Actinopterygii, Chordata). Biodivers Genomes. DOI: https://doi.org/10.56179/001c.56079 DOI: https://doi.org/10.56179/001c.56079
Ryder D, Stone D, Minardi D, Riley A, Avant J, Cross L, Soeffker M, Davidson D, Newman A, Thomson P, et al. 2024. De novo assembly and annotation of the Patagonian toothfish (Dissostichus eleginoides) genome. BMC Genomics 25: 233. DOI: https://doi.org/10.1186/s12864-024-10141-4 DOI: https://doi.org/10.1186/s12864-024-10141-4
Schartl M, Woltering JM, Irisarri I, Du K, Kneitz S, Pippel M, Brown T, Franchini P, Li J, Li M, et al. 2024. The genomes of all lungfish inform on genome expansion and tetrapod evolution. Nature. 634: 96-103. DOI: https://doi.org/10.1038/s41586-024-07830-1 DOI: https://doi.org/10.1038/s41586-024-07830-1
Descargas
Publicado
Número
Sección
Licencia
Derechos de autor 2025 Alejandro S. Mechaly, Yamila P. Cardoso

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial-CompartirIgual 4.0.
Los autores de los artículos publicados en Marine and Fishery Sciences conservan los derechos de autor de sus artículos, a excepción de las imágenes de terceros y otros materiales añadidos por Marine and Fishery Sciences, que están sujetos a los derechos de autor de sus respectivos propietarios. Por lo tanto, los autores son libres de difundir y volver a publicar sus artículos, sujeto a los requisitos de los propietarios de derechos de autor de terceros y sujeto a que la publicación original sea completamente citada. Los visitantes también pueden descargar y reenviar artículos sujetos a los requisitos de citas. La capacidad de copiar, descargar, reenviar o distribuir cualquier material siempre está sujeta a los avisos de derechos de autor que se muestran. Los avisos de copyright deben mostrarse de manera prominente y no pueden borrarse, eliminarse u ocultarse, total o parcialmente. El autoalmacenamiento en servidores y repositorios de preimpresión está permitido para todas las versiones.
Esta revista ofrece a los autores una política de acceso abierto. Los usuarios pueden leer, descargar, copiar, distribuir, imprimir, buscar o vincular los textos completos de los artículos, o usarlos para cualquier otro propósito legal dentro de la licencia Creative Commons 4.0 (BY-NC-SA), sin solicitar permiso previo del editor o del autor. Esto está de acuerdo con la definición BOAI de acceso abierto.






















